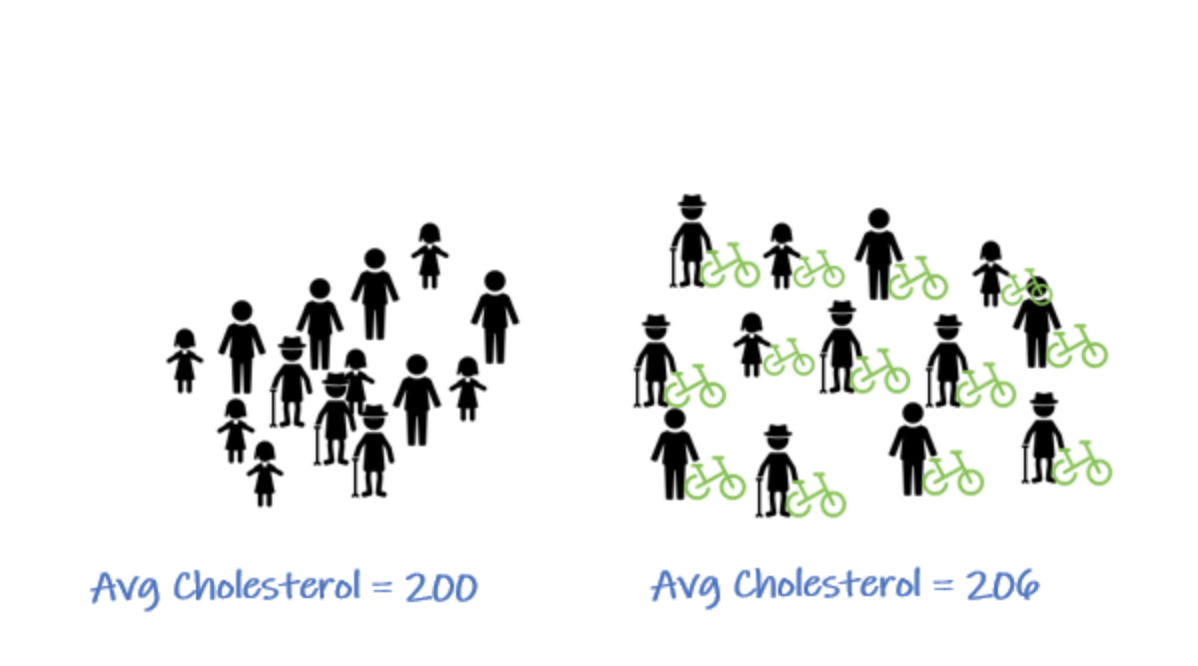
Propensity Score Matching

Last updated


Last updated
# dowhy.causal_estimators.propensity_score_matching_estimator _estimate_effect 함수 코드 일부
from sklearn.neighbors import NearestNeighbors
# ATT (average treatment effect for the treated) 계산
control_neighbors = (
NearestNeighbors(n_neighbors=1, algorithm='ball_tree')
.fit(control['propensity_score'].values.reshape(-1, 1))
)
distances, indices = control_neighbors.kneighbors(treated['propensity_score'].values.reshape(-1, 1))
self.logger.debug("distances:")
self.logger.debug(distances)
att = 0
numtreatedunits = treated.shape[0]
for i in range(numtreatedunits):
treated_outcome = treated.iloc[i][self._outcome_name].item()
control_outcome = control.iloc[indices[i]][self._outcome_name].item()
att += treated_outcome - control_outcome
att /= numtreatedunits
# ATC (average treatment effect for the control) 계산
treated_neighbors = (
NearestNeighbors(n_neighbors=1, algorithm='ball_tree')
.fit(treated['propensity_score'].values.reshape(-1, 1))
)
distances, indices = treated_neighbors.kneighbors(control['propensity_score'].values.reshape(-1, 1))
atc = 0
numcontrolunits = control.shape[0]
for i in range(numcontrolunits):
control_outcome = control.iloc[i][self._outcome_name].item()
treated_outcome = treated.iloc[indices[i]][self._outcome_name].item()
atc += treated_outcome - control_outcome
atc /= numcontrolunits